Both papers, co-led by researcher Ikerbasque Nicola GA Abrescia, have been published in the journal Nature Communications
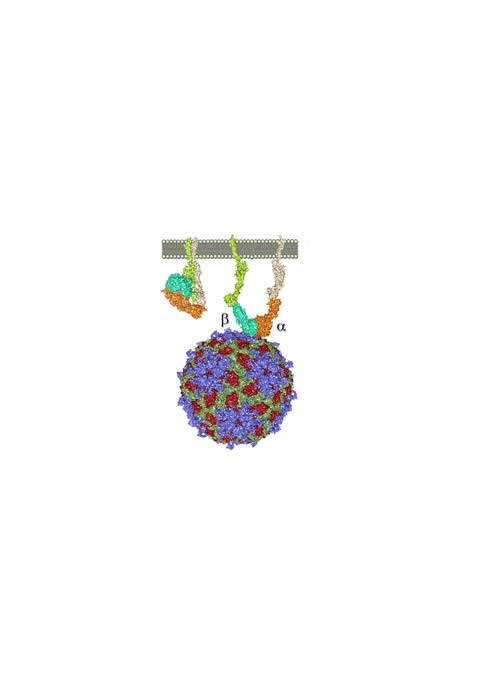
The study on the interaction of foot-and-mouth disease virus with its cellular receptor, integrin alfaVbeta6, will allow the design of inhibitors of virus entry
In both investigations a key role has been played by electron microscopy, fundamental for the compression of cellular mechanisms, including the entry of viruses into cells
Researchers at CIC bioGUNE and the University of Indiana have unveiled the architecture of the archaeal DNA replication machinery assembly, a group of unicellular microorganisms capable of living in extreme conditions, and possibly the oldest forms of life on the earth.
"More than 30 years after their first characterization, we have discovered that PolyB1 DNA polymerase from Sulfolobus is a heterotrimeric holoenzyme. Therefore, assemblages with multi-subunits of the DNA polymerase are found in the three domains of life: eucaria, bacteria and archaea. Our work demonstrates how the association with accessory subunits can affect the central activity of a replicative DNA polymerase, improving its efficiency during genome duplication, a fundamental cellular process in all domains of life, "says Nicola GA Abrescia, Professor Ikerbasque In CIC bioGUNE.
The relevance of this finding, which has been published in the journal Nature Communications, lies in the understanding of the cellular DNA replication mechanism, using archaea as a simplified model system of much more complex eukaryotic processes.
It should be noted that this research may have biotechnological applications, and different companies in this sector are interested in the use of highly stable enzymes, such as DNA polymerases, that can operate in a wide range of working conditions, including temperature and pH .
The project began in 2013 and is the result of collaboration between the laboratories of professors Nicola GA Abrescia at CIC bioGUNE and Stephen Bell at Indiana University. In recent years, this collaboration has led to important contributions in the field of cell transcription and genomic replication.
To carry out this study, in addition to biochemical techniques and X-ray crystallography, electron microscopy methods have been applied. Currently, the use of this technique is allowing spectacular advances in different areas of basic and applied research, including biomedicine.
Research on foot-and-mouth disease virus
Cryo-electron microscopy (cryo-EM) has also been the technique used in other research developed by the CIC bioGUNE team and has also been published in the journal Nature Communications. This study has unraveled the architecture and underlying interactions between foot-and-mouth disease virus (FMDV) and its cellular receptor, integrin alfaVbeta6.
Foot-and-mouth disease virus is a very dangerous pathogen, which has caused enormous losses of animals in the world, with very serious economic consequences. "Understanding the recognition process between foot-and-mouth disease virus and the integrin receptor will open the way for the design of inhibitors of virus entry, and thus prevent or mitigate its spread," explains Nicola GA Abrescia.
Together with CIC bioGUNE, the laboratories of Professor David I Stuart and Juha Huiskonen of Oxford University and Timothy Springer of Harvard University have led this research.
The study on the interaction of foot-and-mouth disease with alfaVbeta6 began in 2005 with collaboration between David I Stuart and Nicola GA Abrescia. The great advances of recent years in the field of cryo-electron microscopy have finally allowed to determine the complete structure of the FMDV-alphaVbeta6 complex and to elucidate the interactions that govern recognition and binding of the virus to the host cell receptor.